Nanopore의 가장 최신 버전인 R9을 기준으로 작성되었습니다.
이전 글에서는 3세대 차세대 염기서열 해독법으로 알려진 나노포어 시퀀싱 기술에 대해 간단히 알아보았습니다. 그렇다면 나노포어 시퀀싱 기술은 nano-meter 사이즈의 포어에 서열을 밀어넣고, 어떻게 이를 측정하고 분석하는 것일까요?
나노포어의 측정/분석 원리를 설명하기 전에 먼저 2019년 기준으로 가장 널리 사용되고 있는 이전 세대의 시퀀서들이 어떻게 염기서열을 측정하는지 간단하게 짚고 넘어가보겠습니다.
다른 장비들의 염기서열 측정 방법
현재 유전체 분석 분야에서 가장 널리 쓰이고 있는 장비는 크게 두 가지로 나뉩니다. 가장 핫하고 분석 방법의 정확도가 높다고 알려진 illumina사의 장비가 있고, 다른 하나는 ThermoFischer Scientific에서 만든 ion Torrent 플랫폼이 있습니다. 재미있게도 이 두 장비 플랫폼은 서로 다른 원리를 사용하여 염기서열을 측정합니다.
illumina 시퀀싱 장비의 원리
illumina 장비에 대한 자세한 설명은 이 영상을 참고하시기 바랍니다.
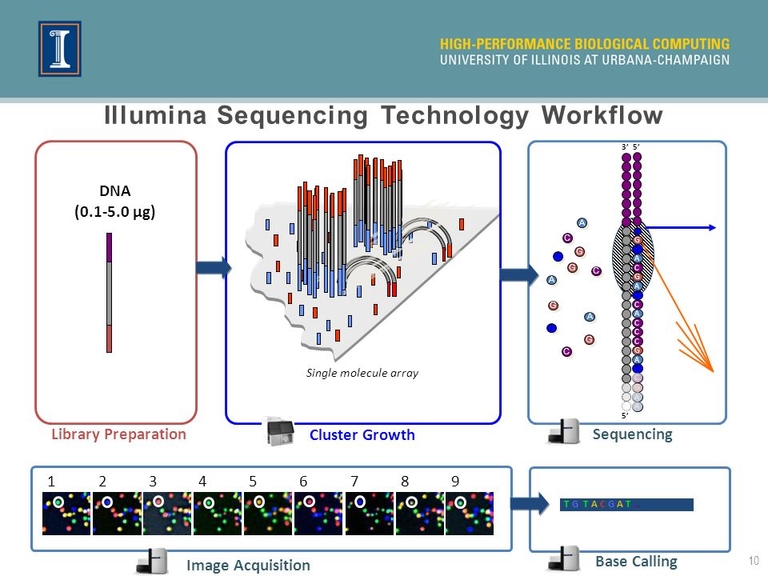
위의 그림과 같이 제작된 DNA 라이브러리는 array의 표면에 붙어 cluster를 형성하게 됩니다. 여기서 중요한 점은 표면에 붙은 라이브러리들은 single strand로 되어있고 DNA Polymerase가 붙어 복제를 할 수 있는 Primer도 존재합니다. 따라서, 우리가 만든 라이브러리 서열들은 Polymerase에 의해 복제되면서 무수히 많은 양의 동일 서열을 만들어냅니다.
하지만, 단순히 복제를 하는 방법으로는 우리가 알고 싶은 정보 즉, 어떤 서열이 존재하는지는 알 수 가 없지요. 그래서 illumina에서는 복제 과정에 꼭 필요한 dNTP (ATCG)에 색이 다른 형광을 부착시켰습니다. 위의 그림에서는 A는 노란색, T는 초록색, C는 빨간색, 그리고 G는 파란색의 형광을 붙인 것으로 보입니다. 색이 다른 이유는 각각의 색이 다른 파장대이기 때문에 후에 측정할 때 구분이 쉽기 때문입니다.
자! 그러면 우리는 이제 dNTP에 형광이 부착되어있기 때문에, Polymerase가 라이브러리 cluster를 복제하는 과정에서 하나씩 서열이 붙으면 여러가지 다른 형광색을 띄겠지요. 측정을 위해서 서열이 붙을 때 마다 레이저를 쏘고, 네 가지 색에 해당하는 파장의 세기를 측정하게 됩니다. 그리고 이때 중요한 점은 형광 값 측정 후에 새로 부착된 서열에서 형광 물질을 떼어내고 남아있는 dNTP들을 씻어 없애줍니다. 이 과정을 통해 하나의 스텝에서는 단 하나의 서열을 정확하게 읽을 수 있게 됩니다. 이렇게 형광 값을 염기서열로 바꿔주는 과정을 Basecalling 이라고 부릅니다.
illumina 플랫폼의 분석 원리는 아주 straightforward 합니다. 단순히 서열이 복제되는 매 스텝마다 사진 이미지를 촬영하고 어떤 파장에서 가장 세기가 강한지를 알아내면 되는 것 입니다. 물론 dNTP가 잘 씻겨나가지 않아 background 신호들이 있을수도 있지만, 이론상으로는 각 스텝에서는 하나의 서열만 있기 때문에 엄청 정확한 염기서열 해독이 가능해진다고 생각합니다.
아마 이러한 이유로 높은 정확도를 가진 illumina 시퀀싱 기술이 현재 가장 대세가 되었던 것이 아닌가 합니다.
Ion Torrent 시퀀싱 장비의 원리

Ion Torrent 장비에 대한 자세한 설명은 이 영상을 참고하시기 바랍니다.
위의 그림과 같이 Ion Torrent 장비는 형광 값으로 서열을 측정하지 않고, 염기서열이 하나씩 복제되는 순간 발생하는 수소 (H) 이온을 측정 하게 됩니다. 우리가 살면서 한 번쯤은 들어본 pH의 값을 측정하게 되면 수소 이온이 생겼는지 안생겼는지 아주 간단하게 알 수가 있습니다. 그리고 이 pH의 변화를 매 스텝 별로 측정하고 pH의 변화에 맞추어 정확하게 염기서열 정보로 다시 변환해주는 기술이 Ion Torrent 시퀀싱 플랫폼의 핵심 기술입니다.
예를 들어 다음 복제될 서열이 C라고 가정해봅시다. 일단 다음 서열이 뭐가 될지 모르니 ATCG dNTP를 각각 한 스텝에 하나씩 넣어 봅니다. 이때, C를 넣는 순간 수소 이온이 발생할 것이고, 이로 인해 기존의 pH에 변화가 생기게 됩니다. 만약 연속으로 C 서열이 두개 필요하면 어떻게 측정되냐구요? 간단합니다! 하나의 C 서열이 붙은 경우 보다 당연히 두 개의 C 서열이 붙은 경우의 pH 변화가 더 크겠지요.
illumina 시퀀싱 플랫폼과 비교했을 때의 장점은 일단 dNTP에 형광을 붙이 필요가 없어 개인적인 생각이지만 시퀀싱 키트 제조 단가가 저렴할 것입니다. 또한, illumina에서는 염기서열을 하나씩 반응시키기 위해 반응이 끝나고 이미지를 촬영한 다음에서야 다시 남아있는 형광 물질과 dNTP를 제거하고, 다시 dNTP를 넣고 반복하게 됩니다. 결론적으로는 step도 많아지고, 측정 시간이 길어집니다. 이와 다르게 Ion Torrent 플랫폼은 dNTP를 한 종류씩 넣어보고 반응시켜 pH를 측정하기 때문에 만약 동일 서열이 반복되는 경우 illumina 장비 보다는 더 빠르게 측정이 가능해집니다.
나노포어는 어떻게 염기서열을 측정할까?
이전 글에서 나노포어 시퀀싱 기술은 나노포어에 단일 가닥의 염기서열이 구멍 내부를 통과하면서 변하게 되는 전류의 값을 측정한다고 정리했었습니다. 어떻게 보면 나노포어 시퀀싱 기술은 Ion Torrent 기술과 약간 비슷한 면이 있습니다. 바로 형광을 측정하는 방식이 아니라, 전기적 신호, 전류나 전압의 변화를 측정한다는 점입니다.
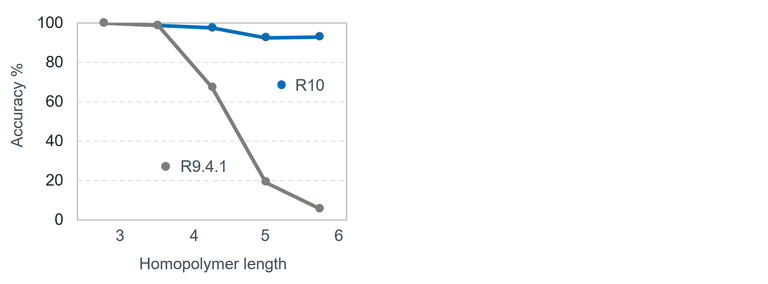
어찌되었건 나노포어는 구멍에 들어가있는 4-5개의 서열을 전류 값을 읽어들입니다. 다른 기술들과 다른 점은 서열을 하나씩 읽지 않는다는 점인데, 이게 양날의 검 같습니다. 하나씩 읽지 않는 방법의 장점은 역시 서열 해독에 걸리는 시간이 줄어든다는 것인데, 문제는 같은 서열이 길게 반복되는 homopolymer 같은 경우 나노포어 플랫폼의 정확도가 많이 떨어진다고 알려져 있습니다.
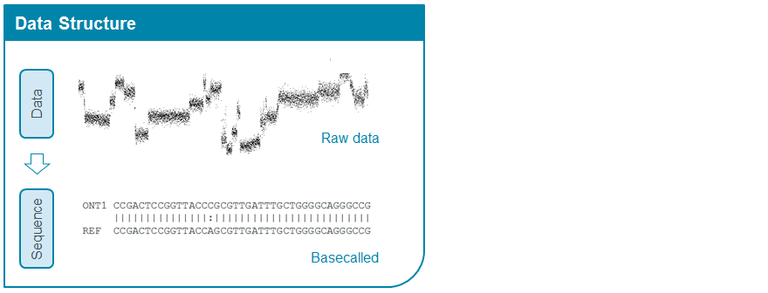
그래도 최근의 분석 기술과 컴퓨팅 기기 성능의 집약적인 발전으로 인해 대량의 데이터로 훈련시킨 인공지능 신경망 사용이 많이 활성화 되었고, 나노포어 플랫폼의 basecalling (전류 신호를 염기서열로 변환) 과정에도 이 인공지능이 들어가있습니다.
ONT사의 공식 홈페이지 글에 따르면, RNN (Recurrent Neural Network)를 사용한다고 나와있습니다. 이 인공신경망은 현재 엄청난 양의 서열이 아주 잘 알려진 Reference 게놈을 시퀀싱한 데이터를 통해 훈련이 된 상태입니다. RNN은 이전에 이미 한 번이라도 본 데이터를 내부 메모리에 저장해놓고 새로운 분석 데이터가 보이면 이를 이전 데이터를 참고하여 분석한다고 합니다. 이 부분은 제가 AI를 좀 더 깊게 공부를 해야 원리를 더 정확하게 설명할 수 있을듯 합니다… 결론적으로는 측정된 전류 신호들은 인공신경망인 RNN을 통해 염기서열로 해독되어 우리에게 FASTQ 파일로 제공됩니다.
현재 ONT사에서 주장하는 나노포어 시퀀싱 플랫폼의 분석 정확도는 약 92-94%정도 라고 합니다.
자료 출처:
[1] A.Magi et al. Briefings in Bioinformatics, 2017, 1-17
[2] ONT official website
[3] Illumina official website
[4] Ion Torrent official website
이번 글에서는 illumina와 Ion Torrent 장비의 염기서열 측정 방법을 알아봄과 동시에 나노포어 플랫폼은 어떻게 염기서열을 해독하는지에 대해 간단히 알아보았습니다.
다음 글에서는 나노포어 시퀀싱 결과 데이터를 가지고 후성유전학 (Epigenetics)중 하나인 Structural Variation을 검출하는 분석 파이프라인을 step by step 짚고 넘어가도록 하겠습니다.
© ChansNotes Powered By Gatsby